Kiindulási anyag: RNS
A kvantitatív reverz transzkripciós PCR (RT-qPCR) egy kísérleti módszer, amelyet a PCR-kísérletekben használnak, RNS-t használva kiindulási anyagként.Ebben a módszerben a teljes RNS-t vagy a hírvivő RNS-t (mRNS) először komplementer DNS-vé (cDNS) írják át a reverz transzkriptáz.Ezt követően a cDNS-t templátként használva qPCR-reakciót végeztünk.Az RT-qPCR-t számos molekuláris biológiai alkalmazásban használták, beleértve a génexpressziós elemzést, az RNS interferencia validálást, a microarray validálást, a kórokozók kimutatását, a genetikai tesztelést és a betegségkutatást.
Egy- és kétlépéses módszerek az RT-qPCR-hez
Az RT-qPCR egy- vagy kétlépéses módszerrel végezhető el.Az egylépéses RT-qPCR egyesíti a reverz transzkripciót és a PCR-amplifikációt, lehetővé téve a reverz transzkriptáz és a DNS-polimeráz számára, hogy ugyanabban a csőben, azonos pufferkörülmények között befejezze a reakciót.Az egylépéses RT-qPCR csak szekvencia-specifikus primerek használatát igényli.A kétlépcsős RT-qPCR során a reverz transzkripciót és a PCR-amplifikációt két csőben hajtják végre, különböző optimalizált pufferek, reakciókörülmények és primer tervezési stratégiák használatával.
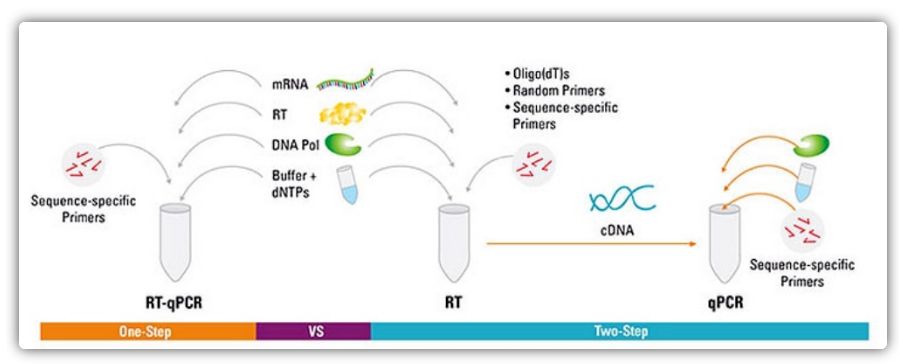
| Előny | Hátrány | |
| Egy lépés | Ez a módszer kevesebb kísérleti hibával jár, mivel mindkét reakciót egy csőben hajtják végre
Kevesebb pipettázási lépés csökkenti a szennyeződés kockázatát
Alkalmas nagy áteresztőképességű erősítésre/szűrőre, gyors és reprodukálható | A kétlépcsős reakciók külön nem optimalizálhatók
Mivel a kétlépéses reakció kombinálásával a reakciókörülmények sérülnek, az érzékenység nem olyan jó, mint a kétlépéses módszeré
Az egyetlen minta által észlelt célpontok száma kicsi |
| Két lépés | Stabil cDNS-könyvtárak létrehozásának képessége, amelyek hosszú ideig tárolhatók és többféle reakcióban felhasználhatók
A célgének és a referenciagének amplifikálhatók ugyanabból a cDNS-könyvtárból anélkül, hogy több cDNS-könyvtárra lenne szükség
Reakciópufferek és reakciókörülmények, amelyek lehetővé teszik az egyes reakciófutások optimalizálását
A trigger feltételek rugalmas kiválasztása | Több cső és több pipettázási lépés használata növeli a DNS-szennyeződés kockázatát, és időigényes.
Több optimalizálást igényel, mint az egylépéses módszer |
Kapcsolódó termékek:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
RT-qPCR Easyᵀᴹ (Egy lépés)-Taqman
RT Easyᵀᴹ I Master Premix Első szálú CDNA-szintézishez
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
A teljes RNS és mRNS kiválasztása
Az RT-qPCR kísérlet megtervezésekor fontos eldönteni, hogy teljes RNS-t vagy tisztított mRNS-t használjunk templátként a reverz transzkripcióhoz.Bár az mRNS valamivel nagyobb érzékenységet biztosíthat, a teljes RNS-t még mindig gyakran használják.Ennek az az oka, hogy a teljes RNS kiindulási anyagként fontosabb előnnyel rendelkezik, mint az mRNS.Először is, a folyamat kevesebb tisztítási lépést igényel, ami biztosítja a templát jobb kvantitatív visszanyerését és az eredmények jobb normalizálását a kiindulási sejtszámhoz.Másodszor, elkerüli az mRNS-dúsítási lépést, amivel elkerülhető a torz eredmények a különböző mRNS-ek eltérő kinyerése miatt.Összességében, mivel a legtöbb alkalmazásban a célgén relatív mennyiségi meghatározása fontosabb, mint a detektálás abszolút érzékenysége, a legtöbb esetben a teljes RNS megfelelőbb.
Reverz transzkripciós primer
A kétlépéses módszerben három különböző módszer használható a cDNS-reakció indítására: oligo(dT) primerek, random primerek vagy szekvencia-specifikus primerek.Jellemzően oligo(dT) primereket és random primereket használnak kombinációban.Ezek a primerek a templát mRNS-szálhoz kapcsolódnak, és reverz transzkriptázt biztosítanak a szintézis kiindulási pontjával.
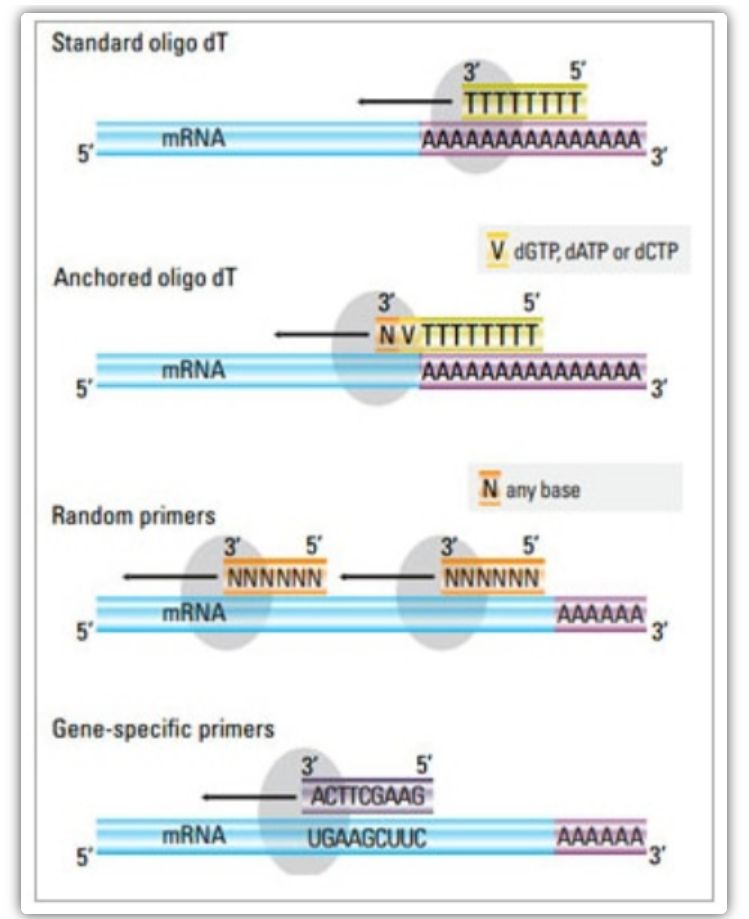
| Primer kiválasztása | Felépítés és funkció | Előny | Hátrány |
| Oligo(dT) primer (vagy lehorgonyzott oligo(dT) primer) | Az mRNS poli(A) farkánál lévő timin-maradékokhoz való kiterjesztett annealing;A horgony oligo(dT) primer G-t, C-t vagy A-t tartalmaz a 3′ végén (horgonyzóhely) | Teljes hosszúságú cDNS szintézise poli(A)-farkú mRNS-ből
Akkor alkalmazható, ha kevesebb kiindulási anyag áll rendelkezésre
A rögzítési hely biztosítja, hogy az oligo(dT) primer kötődjön az mRNS 5′ poli(A) farkához | Csak poli(A)-farokkal rendelkező gének amplifikálására alkalmas
Szerezzen csonkolt cDNS-t a priming helyről*2 a poli(A)-ban
Előfeszítve a 3′ véghez való kötéshez*
* Ez a lehetőség minimálisra csökken, ha rögzített oligo(dT) primereket használnak |
| random primer
| 6-9 bázis hosszúságú, amely az RNS-transzkripció során több helyhez kapcsolódhat | Csatlakozás az összes RNS-hez (tRNS, rRNS és mRNS)
Alkalmas jelentős másodlagos szerkezetű átiratokhoz, vagy ha kevesebb kiindulási anyag áll rendelkezésre
Magas cDNS-hozam | A cDNS az összes RNS-ről reverz átírásra kerül, ami általában nem kívánatos, és felhígíthatja a cél-mRNS jelét
csonkolt cDNS-t kap |
| szekvencia-specifikus primerek | Egyedi primerek, amelyek specifikus mRNS-szekvenciákat céloznak meg | specifikus cDNS könyvtár
Az érzékenység javítása
Fordított qPCR primerek használata | Csak egyetlen célgén szintézisére korlátozódik |
Reverz transzkriptáz
A reverz transzkriptáz egy olyan enzim, amely RNS-t használ a DNS szintetizálására.Egyes reverz transzkriptázok RNáz aktivitással rendelkeznek, és a transzkripció után lebonthatják az RNS-DNS hibrid szálak RNS-szálait.Ha nem rendelkezik RNáz enzimaktivitással, RNázH hozzáadható a nagyobb qPCR hatékonyság érdekében.Az általánosan használt enzimek közé tartozik a Moloney egér leukémia vírus reverz transzkriptáza és a madár myeloblastoma vírus reverz transzkriptáza.Az RT-qPCR-hez ideális nagyobb termostabilitású reverz transzkriptázt választani, így a cDNS szintézis magasabb hőmérsékleten is végrehajtható, biztosítva a magasabb másodlagos szerkezetű RNS-ek sikeres transzkripcióját, miközben a reakció teljes időtartama alatt megőrzi teljes aktivitásukat, ami magasabb cDNS hozamot eredményez.
Kapcsolódó termékek:
Foreasy M-MLV reverz transzkriptáz
A reverz transzkriptáz RNáz H aktivitása
Az RNaseH képes RNS-szálakat lebontani az RNS-DNS duplexekből, lehetővé téve a kettős szálú DNS hatékony szintézisét.Ha azonban hosszú mRNS-t használunk templátként, az RNS idő előtt lebomolhat, ami csonkolt cDNS-t eredményez.Ezért gyakran előnyös az RNázH aktivitás minimalizálása a cDNS klónozás során, ha hosszú transzkriptumok szintézisére van szükség.Ezzel szemben az RNáz H aktivitással rendelkező reverz transzkriptázok gyakran előnyösek a qPCR alkalmazásokban, mivel fokozzák az RNS-DNS duplexek olvadását a PCR első ciklusában.
Primer design
Az RT-qPCR-ben a qPCR lépéshez használt PCR primereket ideális esetben úgy kell megtervezni, hogy egy exon-exon csomópontot fedjenek le, ahol az amplifikációs primer potenciálisan átnyúlhat egy tényleges exon-intron határon.Mivel az intront tartalmazó genomiális DNS-szekvenciák nem amplifikálódnak, ez a kialakítás csökkenti annak a kockázatát, hogy a genomiális DNS-t szennyező álpozitívek amplifikálódjanak.
Ha a primereket nem lehet megtervezni az exonok vagy az exon-exon határok elválasztására, szükséges lehet az RNS-mintákat RNáz-mentes DNáz I-gyel vagy dsDNázzal kezelni a genomi DNS-szennyeződés eltávolítása érdekében.
RT-qPCR vezérlés
Minden RT-qPCR-kísérletben reverz transzkripciós negatív kontrollt (-RT-kontrollt) kell tartalmazni a DNS-szennyeződés (például genomi DNS vagy korábbi reakciókból származó PCR-termékek) kimutatására.Ez a kontroll a reverz transzkriptáz kivételével az összes reakciókomponenst tartalmazza.Mivel reverz transzkripció nem fordul elő ezzel a kontrollal, ha PCR-amplifikációt észlelünk, a DNS-ből való szennyeződés a legvalószínűbb.
Feladás időpontja: 2022-02-02








